Die flexible Lösung für SNP Genotyping im Hochdurchsatz
NEBNext Direct Genotyping Solution (GS) ist die neue flexible und kostengünstige Methode für Genotyping by Sequencing / SNP Genotyping im Hochdurchsatz auf Illumina Plattformen.
Unsere Schlüsseltechnologie dabei ist die hybridisierungsbasierte NEBNext Direct Target Enrichment Methode. Sie ist besonders effizient und wurde daher mit dem “Application Award 2020” im Bereich Bio- und Pharmaanalytik im Rahmen der Analytica virtual ausgezeichnet wurde.
NEBNext Direct Genotyping Solution wird in verschiedenen Anwendungen erfolgreich eingesetzt:
 |
 |
 |
| Pflanzenzüchtung | Tierzucht | Humanmedizin |
| Marker Assisted Selection/ Breeding | Genotypisierung | Biobanking |
| QTL Screening | Viehzucht | NGS Sample Tracking |
Hybridierungsbasiertes Target Enrichment – So einfach funktioniert‘s:
Sie übermitteln uns die für Ihre Anwendung und Ihr Probenmaterial/Spezies relevanten 100 – 5.000 Marker bzw. SNPs. Wir von NEB entwickeln und optimieren für Sie komplementäre Baits zur hochspezifischen Zielanreicherung mit der NEBNext Direct Target Enrichment Technologie. Zur Qualitätssicherung stellen Sie uns dazu die entsprechende DNA aus Ihrem Probenmaterial zur Verfügung. So entstehen die perfekten Baits – getestet, spezifisch und optimiert für Ihre Anwendung!
Schauen Sie das Webinar:
“Fully automated targeted SNP genotyping for plant and animal applications”
Der „Application Award 2020“ für NEBs Innovation und Effizienz im Bereich SNP Genotyping im Hochdurchsatz (mehr…).
Workflow:
In einem Pre-Capture Multiplexing Schritt kombinieren Sie bis zu 96 Proben. Nach der Zielanreicherung mit der NEBNext Direct Target Enrichment Technologie wird dieser Sample-Pool mit einem von 8 Standard-Pool-Indizes versehen. Durch Vereinigung dieser Pools können Sie so bis zu 768 Proben bzw. 3,8 Millionen Genotypen in einem einzigen Illumina-Lauf sequenzieren und analysieren.
Auf Anfrage stehen Ihnen für erweitertes Multiplexing sogar weitere Pool-Indizes zur Prozessierung von bis zu 9.216 Proben in einem Illumina Sequenzier-Lauf zur Verfügung!
Und das sagen unsere Kunden:
|
„For our daily work, we needed a technology that stands out in robustness, quality of data, multiplexing level, flexibility and throughput. We found all of that in the NEBNext Direct Genotyping Solution.“ Senior Molecular Biology Researcher at Enza Zaden |
||||
 |
||||
|
„We had the opportunity to test the NEBNext Direct Genotyping Solution in order to assess the feasibility for marker-assisted breeding in rice. The technology delivered excellent data and NEB delivered exceptional support and follow-up“ Embrapa Bean and Rice |
||||
 |
||||
Interesse?
Dann kontaktieren Sie unsere NEBNext Direct Genotyping Solutions Experten per Webformular oder per E-Mail an nebnextdirect@neb.com .
Gerne stehen Ihnen auch unsere Kollegen und Kolleginnen vom technischen Support zur Verfügung.
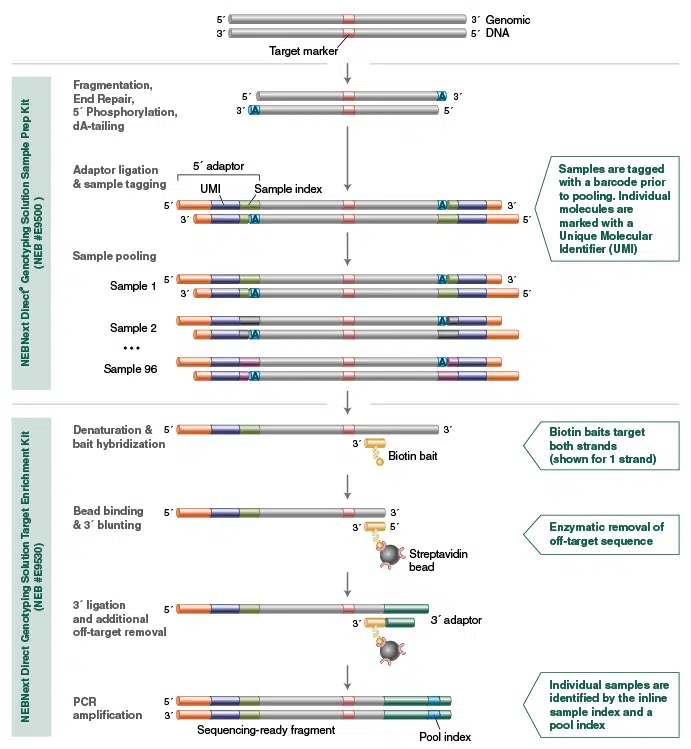
Der NEBNext Direct Genotyping Solution Workflow
Als Input für den NEBNext Direct Genotyping by Sequencing Workflow werden 25 – 100 ng aufgereinigte genomische DNA benötigt. Die DNA wird enzymatisch fragmentiert und an Illumina-kompatible P5-Adaptoren ligiert. Die Adaptoren enthalten einen individuellen Sample-Index sowie einen Unique Molecular Identifier (UMI) für die Identifizierung von Duplikaten.
Bis zu 96 Samples können vor dem Hybridisierungs-basierten Target Enrichment Schritt gepoolt werden. Für das Target Enrichment werden biotinylierte Baits verwendet, die mit Streptavidin Beads angereichert werden. Anschließend werden die Off-Target Sequenzen entfernt und die Libraries mittels PCR amplifiziert. Dabei wird ein 3′ Pool Index in die Libraries eingefügt, welcher für jeden 96-Sample Pool spezifisch ist. Dadurch können mehrere Pools in einem Lauf sequenziert werden. So wird die Kapazität des Sequenziers optimal ausgelastet und die Sequenzierkosten werden minimiert.
- Zeitersparnis durch einen schnellen Workflow: Vom Sample zur Library in nur einem Tag.
- 96-plex Pre-Capture Sample Multiplexing von 100 - 5.000 Markern
- Prozessierung von bis zu 9.160 Proben in einem einzigen Sequenzier-Lauf
- Bait-Design und Sample Multiplexing für effiziente Sequenzierer-Auslastung
- Hohe Spezifität und gleichmäßige Coverage
Performance
Gleichmäßige Coverage
Das Hybridisierungs-basierte Target-Enrichment liefert im Gegensatz zu PCR Enrichment Methoden eine sehr gleichmäßige Coverage der angereicherten DNA Bereiche. Die einzigartige enzymatische Entfernung von Off-Target Regionen erhöht die Spezifität zusätzlich. Dadurch reduziert sich die Zahl der notwendigen Sequenzierreads pro Target und mehr Targets können in einer Analyse zusammengefasst werden.
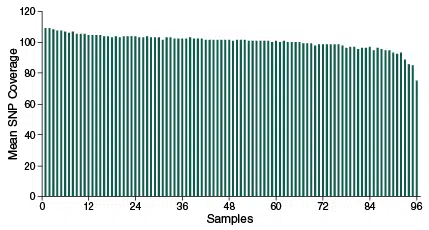
Gleichmäßige Coverage bei 96 Proben
Mittlere Abdeckung von 2.309 SolCAP-Markern bei 96 Proben in einer SNP Genotyping Analyse. Für jede Probe wurden 25 ng aufgereinigte Tomaten-DNA verwendet. Die Proben wurden vor der Hybridisierung mit einem Index versehen und gepoolt. Die Libraries wurden auf einem Illumina MiSeq mit 20 Zyklen von Read 1 (UMI- und Index-Read) sowie 75 Zyklen von Read 2 (Target-Read) sequenziert.
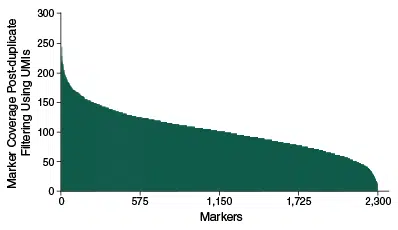
Gleichmäßige Coverage über 2309 Marker
Das Histogramm zeigt die gleichmäßige Abdeckung von 2.309 SolCAP-Markern innerhalb eines Samples aus einem 96 Sample-Pool. Für jede Probe wurden 25 ng aufgereinigte Tomaten-DNA verwendet. Die Proben wurden vor der Hybridisierung mit einem Index versehen und gepoolt. Die Libraries wurden auf einem Illumina MiSeq mit 20 Zyklen von Read 1 (UMI- und Index-Read) sowie 75 Zyklen von Read 2 (Target-Read) sequenziert.
Spezifisches Target Enrichment
Das Pre-Capture-Pooling von 96 Proben bietet verschiedene Vorteile. Zum einen werden die erforderlichen Pipettierschritte und damit die “Hands-On-Time” erheblich reduziert, es werden weniger Verbrauchsmaterialien benötigt und so Kosten und Umweltbelastung gesenkt.
Da bis zu 9216 Samples in einem einzigen Sequenzierlauf kombiniert werden können, werden Sequenzierkapazitäten optimal ausgelastet und so ein höherer Durchsatz bei geringeren Kosten ermöglicht.
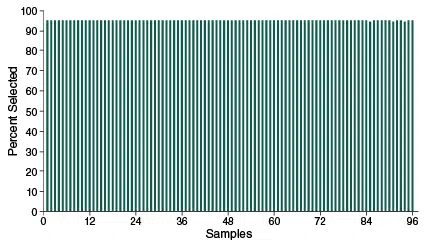
Spezifisches Enrichment 96 Proben
Der Prozentsatz gültiger Reads zeigt die hohe Spezifität auch beim Poolen von 96 Proben. 25 ng aufgereinigte Tomaten-DNA wurden jeweils als Input verwendet. Die Proben wurden vor der Hybridisierung mit einem Index versehen und gepoolt. Die Libraries wurden auf einem Illumina MiSeq mit 20 Zyklen von Read 1 (UMI- und Index-Read) sowie 75 Zyklen von Read 2 (Target-Read) sequenziert.
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.



