Sehen Sie eine realistische Abbildung
Ihrer small RNAs
Eine verfälschte Repräsentation, insbesondere durch seltene RNA-Spezies bei der Small-RNA Library Prep können die Genauigkeit Ihrer Daten erheblich beeinträchtigen. Das NEBNext Low-bias Small RNA Library Prep Kit minimiert diese Verzerrungen und sorgt für eine realistischere Abbildung der Anzahl und Verteilung einzelner Small RNAs.
Der Workflow ist auf die Erfassung von Small RNAs (<120 Nukleotide) mit einer 5′-Phosphat- und 3′-Hydroxylgruppe optimiert und eignet sich zur Sequenzierung von microRNAs (miRNAs), Transfer-RNAs (tRNAs) und tRNA-Fragmenten, small nucleolar RNAs (snoRNAs), piwi-interagierenden RNAs (piRNAs) sowie pflanzlichen miRNAs.
Das Kit wurde speziell für die Erstellung von Illumina-kompatiblen small-RNA Libraries entwickelt.
- Analyse der RNA-Spezies, ohne Verzerrungen
- Einfache und schnelle Library Prep – in nur 3,5 Stunden
- Breite, biologisch relevante Inputmengen
- Einheitliches Protokoll für Standardproben und 2'-O-methylierte RNA
- 480 kompatible UDI-Primerpaare separat erhältlich
Fordern Sie jetzt ein Sample kostenfrei an:
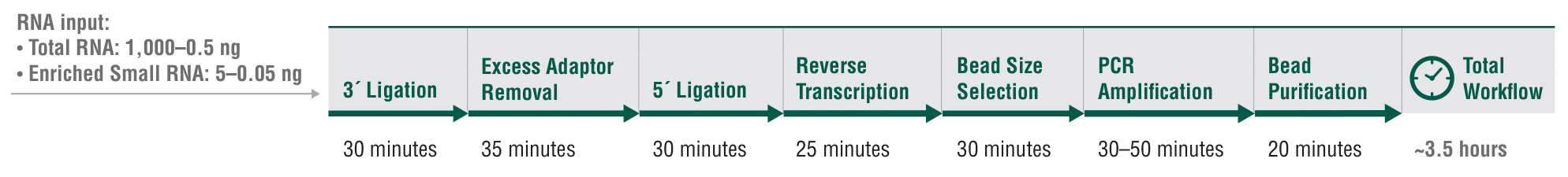
Der schnelle NEBNext Low-bias Small RNA Library Prep Workflow:
Das NEBNext Low-bias Small RNA Library Prep Kit produziert im Vergleich die Libraries mit der realistischsten Repräsentation der verschiedenen small RNAs:
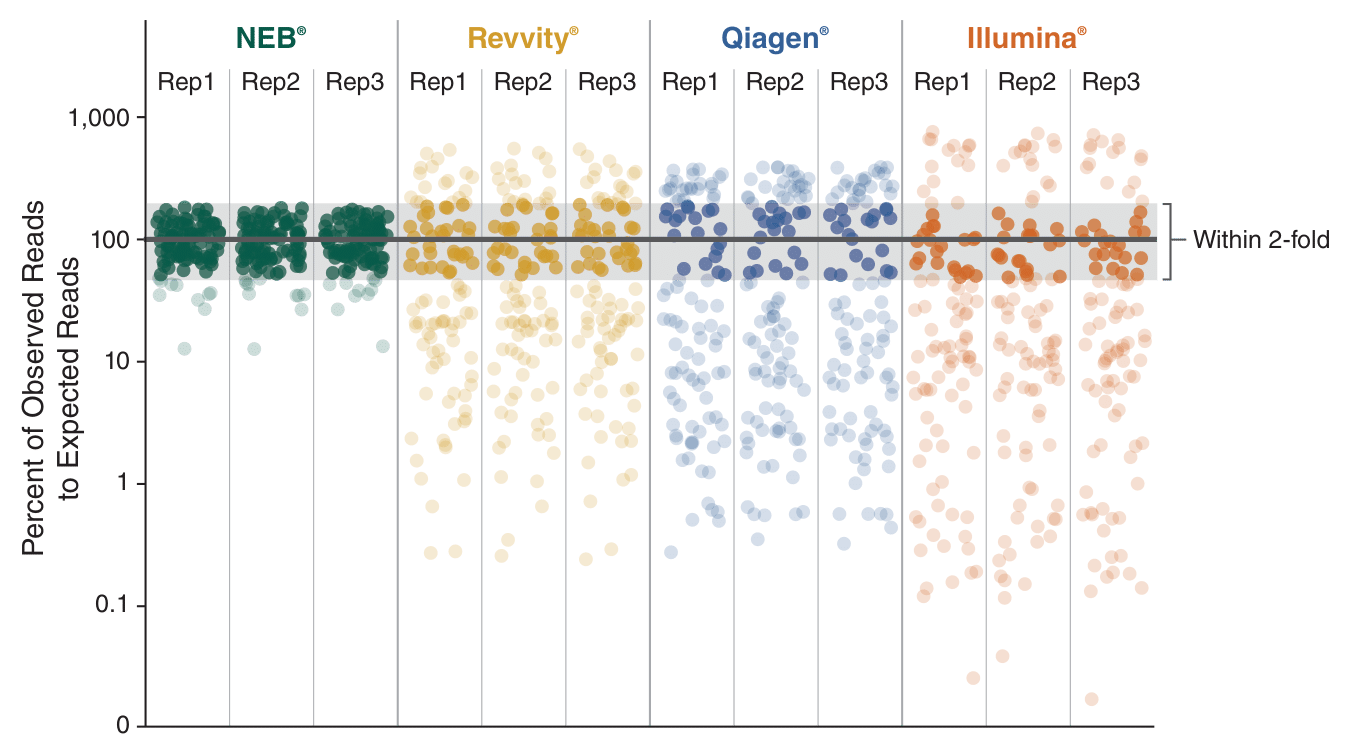
Small RNA-Libraries wurden aus 0,3 ng eines Mixes aus 100 synthetischen Kontroll-miRNAs erstellt, darunter fünf mit 3′-2′-O-Methyl-Enden. Um die Verzerrung durch die Library Prep zu vergleichen, wurden Libraries mit dem NEBNext Low-bias Small RNA Library Prep Kit und Kits von Revvity (NEXTFLEX Small RNA Sequencing Kit V4), Qiagen (QIAseq miRNA Library Kit) und Illumina (TruSeq Small RNA Library Prep) erstellt. Die Sequenzierung erfolgte auf einem Illumina NextSeq 500 (1 × 56 Basen). Die erwarteten Reads wurden berechnet, indem die Gesamtzahl der auf die synthetischen Kontrollen gemappten Reads durch die Anzahl der Kontrollsequenzen geteilt wurde (Referenzlinie bei 100 %). Der Prozentsatz der beobachteten zu den erwarteten Reads wurde für jede Kontrollsequenz berechnet und über Replikate hinweg dargestellt.
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.


