Nutzen Sie für all Ihre Restriktionsverdaus die High Fidelity (HF®) Restriktionsenzyme!
Ihre Vorteile
 Engineered for performance
Engineered for performance
New England Biolabs bietet Ihnen die besten Restriktionsenzyme aller Zeiten! Durch den gezielten Austausch von Aminosäuren hat New England Biolabs eine einzigartige neue Generation an sogenannten High Fidelity (HF®) Restriktionsenzymen konstruiert, die Ihnen als Anwender viele Vorteile bieten! Die HF-Enzyme sind einzigartige, funktionsoptimierte Designer-Enzyme.
 Erhöhte Genauigkeit dank dramatisch reduzierter Star Aktivität
Erhöhte Genauigkeit dank dramatisch reduzierter Star Aktivität
NEBs HF-Enzyme (z.B. EcoRI-HF®) erkennen die gleichen definierten Schneidesequenzen auf ihrer Substrat-DNA wie ihre herkömmlichen „Wildtyp”-Pendants. Durch die dramatische Reduktion der intrinsichen Star-Aktivität („off-target“ Aktivität und Abbau von DNA-Enden) bei allen HF-Enzymen ist die Genauigkeit des DNA-Verdaus um bis zu 62.500-fach erhöht! Gelegentlich auftretende Probleme beim Klonieren mit Wildtyp-Enzymen wie z.B. EcoRI, BamHI oder SalI gehören somit der Vergangenheit an.
Nutzen Sie für Ihre Klonierungen, Genotypisierungen etc. die neuen HF-Enzyme.
Klicken Sie hier für weitere Details.

 Optimiert für höchste Puffer-Kompatibilität (NEBCutSmart)
Optimiert für höchste Puffer-Kompatibilität (NEBCutSmart)
Um die bestmögliche Kompatibilität des Reaktionspuffers zu erreichen, sind alle HF-Enzyme optimiert für 100% Aktivität im rCutSmart Puffer. Dieser Puffer garantiert für über 200 NEB Enzyme inkl. aller HF-Enzyme eine 100%ige Aktivität im Einzel- sowie Doppelverdau – ganz ohne Star-Aktivität – und vereinfacht somit Ihren Reaktionsansatz. Nie waren Doppelverdaus einfacher als heute. HF-Enzyme sind vollständig kompatibel mit den Standard NEB „Wildtyp“ Enzymen. Mit den High Fidelity Restriktionsenzymen ist der DNA-Verdau nicht nur sicherer geworden in punkto Star-Aktivität, sondern auch einfacher in der Anwendung.
 Time-Saver™ qualifiziert für optionale 15 min Verdaus
Time-Saver™ qualifiziert für optionale 15 min Verdaus
Alle HF-Enyme sind „Time-Saver™ qualifiziert“(![]() ), d.h. sie schneiden Ihre Substrat-DNA (Lambda oder Plasmid) in nur 15 min! Ob Sie nun die HF-Enzyme tatsächlich für einen schnellen „Time-Saver™“-Verdau in nur 15 min nutzen oder wie gewohnt länger inkubieren möchten (z.B. in einem Übernachtverdau), entscheiden Sie selbst: Auch bei verlängerten Inkubationszeiten oder erhöhten Enzymkonzentrationen bleibt Ihre Ziel-DNA vollständig intakt und die Enden ligierbar! Das HF-Enzym „richtet sich“ nach Ihren Vorlieben und Ihren Dienstplan.
), d.h. sie schneiden Ihre Substrat-DNA (Lambda oder Plasmid) in nur 15 min! Ob Sie nun die HF-Enzyme tatsächlich für einen schnellen „Time-Saver™“-Verdau in nur 15 min nutzen oder wie gewohnt länger inkubieren möchten (z.B. in einem Übernachtverdau), entscheiden Sie selbst: Auch bei verlängerten Inkubationszeiten oder erhöhten Enzymkonzentrationen bleibt Ihre Ziel-DNA vollständig intakt und die Enden ligierbar! Das HF-Enzym „richtet sich“ nach Ihren Vorlieben und Ihren Dienstplan.
 Günstiger Preis
Günstiger Preis
NEB ist Ihr fairer Partner!
Wir möchten, dass Sie mit den bestmöglichen „Werkzeugen“, d.h. unseren HF-Restriktionsenzymen arbeiten und damit erfolgreich sind! Daher zahlen Sie für die HF-Enzyme keine Mehrkosten, sondern profitieren vom gewohnt günstigen NEB Preis! (![]() ).
).
Unsere uneingeschränkte Empfehlung lautet: Nutzen Sie für Ihre DNA Experimente stets die verbesserten High Fidelity (HF®) Restriktionsenzyme!
![]()
Inkl. kostenfreiem Bonus: 1 ml Gel Loading Dye, Purple (6X) gratis
Erhältliche HF-Enzyme
|
|
|
HF-Enzyme und reduzierte Star-Aktivität
Als Star-Aktivität bezeichnet man die intrinsische Eigenschaft vieler Restriktionsenzyme, neben der definierten „kanonischen“ Erkennungssequenz (z.B. GAATTC) zusätzlich auch ähnliche Nukleotidfolgen (z.B. NAATTC) zu erkennen und zu schneiden („off-target“ Aktivität). Dieses Phänomen tritt insbesondere unter suboptimalen Versuchsbedingungen auf (z.B. pH, Ionenstärke, Verunreinigungen…). Zusätzliche unspezifische Schnitte können nicht nur die Interpretation von Daten erschweren, sondern auch zu falschen Ergebnissen führen, z.B. bei Genotypisierungen oder Klonierungen. Um Star-Aktivität mit Wildtyp-Restriktionsenzymen zu vermeiden, sind diverse vorbeugende Maßnahmen beim Versuchsansatz zu treffen (siehe Tipps und Tricks).
Einfacher geht es mit den HF-Enzymen von New England Biolabs. Durch gezielte Aminosäureaustausche haben wir die Star-Aktivität der HF-Enzyme dramatisch reduziert. So erhalten Sie mit den HF-Restriktionsenzymen eine bis zu 62.500 fach höhere Genauigkeit Ihres Verdaus. Mit HF®-Restriktionsenzymen nehmen Sie das Risiko falscher Klonierungen oder anderer falscher Ergebnisse durch Star-Aktivität aus Ihren Experimenten.
In der folgenden Tabelle finden Sie eine Übersicht aller HF®-Faktoren, dem Mass für reduzierte Star-Aktivität im Vergleich zum entsprechenden herkömmlichen „Wildtyp“ Enzym:
| Enzym | HF Faktor |
| AgeI-HF® | 8 |
| BamHI-HF® | 125 |
| BmtI-HF® | 62500 |
| BsaI-HF® | 250 |
| BstEII-HF® | 125 |
| DraIII-HF® | 1.000 |
| EagI-HF® | 2 |
| EcoRI-HF® | 64 |
| EcoRV-HF® | 64 |
| HindIII-HF® | 2000 |
| KpnI-HF® | 62.500 |
| MfeI-HF® | 16 |
| NcoI-HF® | 530 |
| NheI-HF® | 266 |
| NotI-HF® | 16 |
| PstI-HF® | 33 |
| PvuI-HF® | 32 |
| PvuII-HF® | 32 |
| SacI-HF® | 266 |
| SalI-HF® | 8.000 |
| SbfI-HF® | 32 |
| ScaI-HF® | 62 |
| SphI-HF® | 250 |
| SspI-HF® | 16 |
| StyI-HF® | 125 |
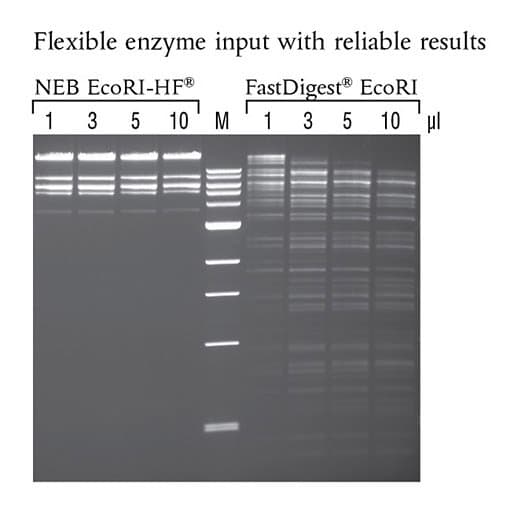
EcoRI-HF® (NEB #R3101) zeigt keine Star-Aktivität, selbst in kritischen Anwendungen wie Übernachtverdau mit hohen Enzymkonzentrationen. Es wurden in 50 µl Reaktionen 1 µg Lambda DNA mit den angegebenen Mengen des jeweiligen Enzyms über Nacht bei 37°C in dem jeweils mitgelieferten optimalen Reaktionspuffer inkubiert. Marker M ist die 1 kb DNA Ladder (NEB# N3232).
HF® is a registered trademark of New England Biolabs, Inc.
FastDigest® is a registered trademark of ThermoScientific (Fermentas).
Tipps & Tricks
| Bedingungen, die Star-Aktivität begünstigen | Vorbeugende Maßnahmen |
| Hohe Glyzerin-Konzentrationen (>5% v/v) | Restriktionsenzyme sind in 50% Glyzerin gelagert. Somit sollte der Anteil des Enzymes im Gesamt- Ansatz 10% nicht überschreiten! Beispiel: in einem 10 µl Gesamt-Ansatz niemals mehr als 1 µl Enzym verwenden! Achtung: kleine Volumina evaporieren leicht und tragen somit zur erhöhten Glyzerinkonzentration bei: hier droht Star-Aktivität! Unser Tipp: Nutzen Sie lieber 50 µl Gesamt-Ansätze! So fallen Evaporation sowie Pipettierungenauigkeiten weniger ins Gewicht und die Enzymmenge kann bei Bedarf ohne Risiko erhöht werden. |
| Große Enzymkonzentrationen (Verhältnis Unit-Menge/µg DNA) |
Nutzen Sie möglichst wenig Enzym für Ihren Verdau. Im Idealfall fahren Sie eine kleine Verdünnungsreihe und „tasten“ sich so an die optimale Enzymmenge heran. Achtung: Die optimale Enzymmenge kann von DNA zu DNA variieren und ist z.B. abhängig von der Struktur, Sequenz und dem Verhältnis der Anzahl an Schnittstellen zur Gesamtlänge der Substrat-DNA. |
| Sub-optimale Pufferbedingungen | Wenn möglich, nutzen Sie immer den mitgelieferten, optimalen NEBuffer. Mit anderen Puffern, insb. bei anderen Ionenstärken und pH-Werten, droht Star-Aktivität! |
| Substitution von Mg2+ Ionen durch andere divalente Kationen (Mn2+, Cu2+, Co2+, Zn2+) | Wenn Sie tatsächlich einen eigenen Puffer verwenden müssen, nutzen Sie unbedingt Mg2+ Ionen als divalente Kationen. Andere divalente Kationen passen häufig nicht zum aktiven Zentrum des Enzyms und interferieren daher mit der korrekten Erkennung der Bindesequenz: hier droht Star-Aktivität! |
| Verlängerte Inkubationen | Nutzen Sie möglichst kurze Inkubationszeiten und stoppen/ beenden Sie die Reaktion bei vollständigem Verdau! Längere Inkubationszeiten – insb. Übernacht-Verdaus – begünstigen Star-Aktivität. |
| Gegenwart von organischen Lösungsmitteln (DMSO, Ethanol, Ethylenglykol, Dimethylacetamid, Dimethylformamid…) | Stellen Sie sicher, dass keine der genannte Lösungsmittel Ihre DNA-Präparation verunreinigen (z.B. als Präparationsrückstände). |
Referenzen:
Nasri M. and Thomas D. (1987) Nucleic Acids Res., 15, 7677.
Malyguine E. and Vannier P. and Yot. (1980) Gene, 8, 163.4.
Verdone L. et al. (1996) Mol. and Cell. Biol., 16, 1978-1988.
Wei, H. et al (2008) Nucleic Acids Reseach 36, e50
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.

